Microscopia de Fluorescência na Pesquisa Biológica: Desafios e uma Nova Solução
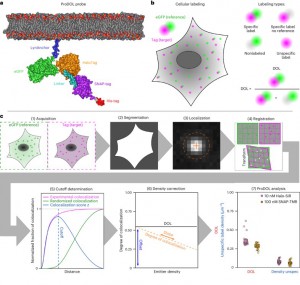
Legenda: a, Princípio e Fluxo de Trabalho do ProDOL. a. Modelo molecular da sonda ProDOL gerado usando AlphaFold com modificação pós-predição, utilizando estruturas publicadas de componentes individuais (Protein Data Bank 2B3Q, 6Y8P e 6U32) e uma bicamada lipídica modelada. b. Modelo esquemático para determinar o DOL (grau de rotulagem) com base na análise de colocalização de moléculas individuais entre uma referência eGFP e o rótulo alvo. Na análise ProDOL, quatro tipos de rotulagem podem ser discernidos (rótulo específico, rótulo específico sem referência, não rotulado e rótulo não específico), e usados para calcular o DOL e a densidade de rotulagem não específica. c. Fluxo de trabalho da análise ProDOL. (1) Imagens adquiridas com ambos os canais de referência e alvo são usadas como entrada. (2) O canal de referência é usado para gerar uma máscara celular. (3) As localizações dos sinais de referência e alvo são ajustadas com precisão subpixel. (4) Os canais são alinhados usando registro afim dos dados de localização. (5) Cálculo do limite de colocalização T. (6) Fatores são determinados para corrigir o DOL para a densidade do emissor. (7) O DOL e a densidade de rótulo não específico (Density unspec) são determinados para todos os canais de tag-alvo adquiridos. Os gráficos de caixa abrangem o intervalo do percentil 25 ao 75, com a mediana indicada por uma linha horizontal dentro da caixa. As linhas de erro se estendem até 1,5 vezes o intervalo interquartil.
Creditos da imagem: Tashev et al, 2024
A microscopia de fluorescência tem sido uma ferramenta essencial na pesquisa biológica, permitindo a detecção de proteínas de interesse (POIs) em uma variedade de contextos. No entanto, a medição confiável do número de cópias de uma POI com essa técnica requer não apenas uma metodologia para contar fluoróforos, mas também informações sobre a fração da POI marcada com marcadores fluorescentes. Técnicas de marcação por afinidade, como a imunomarcação, resultam em eficiências de marcação variáveis e difíceis de caracterizar. Por outro lado, embora a fusão genética com proteínas fluorescentes possa produzir uma proporção de um para um entre o marcador e a POI, ela muitas vezes não é adequada para medições quantitativas devido à fotostabilidade insuficiente e estados de brilho mal definidos. Além disso, proteínas fluorescentes em constructos de fusão podem apresentar uma eficiência aparente de marcação mais baixa devido a eficiências de maturação variáveis ou à formação ineficiente de cromóforos em diferentes ambientes subcelulares. Etiquetas proteicas autolabeláveis, como SNAP-tag e HaloTag, que ligam no máximo um marcador por etiqueta, também são fundidas geneticamente à POI e podem se ligar a uma variedade de substratos fluorescentes com propriedades fotofísicas potencialmente superiores. No entanto, a etapa adicional de marcação que requer incubação com substratos fluorescentes pode resultar em eficiências de marcação variáveis, dependendo da condição escolhida. Além disso, a ligação inespecífica desses substratos na amostra pode resultar em sinais inespecíficos, afetando análises quantitativas ou de colocalização subsequentes.
Para garantir condições ideais de marcação para a técnica de imagem específica, é necessário determinar o grau de marcação (DOL), ou a proporção de marcadores fluorescentes para POI. Um DOL determinado com precisão pode servir como fator de correção para os números de cópias de proteínas medidas em complexos e para as concentrações de proteínas obtidas a partir de técnicas de microscopia de fluorescência. No entanto, a determinação do DOL pode ser desafiadora, e diferentes métodos foram desenvolvidos para abordar essa questão. Uma abordagem comum baseia-se em padrões de contagem molecular, como o complexo do poro nuclear (NPC), combinado com métodos de contagem de fluoróforos, como eficiência de marcação eficaz baseada em super-resolução (ELE), análise rápida de etapas de fotobleaching (quickPBSA) ou contagem por estatísticas de fótons (CoPS). No entanto, o uso de complexos proteicos com estequiometria conhecida muitas vezes traz limitações substanciais, pois a montagem completa do complexo deve ser garantida, e é necessário o uso de uma linha celular knockin homozigótica. Além disso, métodos como ELE e CoPS requerem o uso de marcadores adequados para microscopia de super-resolução ou instrumentação especializada, limitando ainda mais a aplicação geral.
A análise de colocalização com uma etiqueta adicional, espectralmente diferente, em proximidade espacial próxima foi proposta anteriormente para estimar o DOL de etiquetas usando fusões como SNAP-tag e HaloTag. Outros trabalhos utilizaram uma marcação adicional por anticorpos contra as etiquetas proteicas. No entanto, todos os métodos propostos podem sofrer com a marcação inespecífica do sinal de referência, resultando em subestimação do DOL.
Para superar essas limitações, foi proposta uma sonda de calibração modular de DOL que emprega uma proteína fluorescente como sinal de referência quase livre de fundo, combinada com etiquetas proteicas. Este constructo pode ser expresso de forma transitória ou estável em várias linhas celulares, fornecendo uma maneira de medir a eficiência da marcação através da colocalização a nível de molécula única, aumentando a confiabilidade e a versatilidade das medições. Na implementação atual, a sonda de calibração de DOL é composta por uma proteína verde fluorescente aprimorada ancorada na membrana (eGFP) fundida a uma SNAP-tag e HaloTag, chamada de sonda “protein-tag degree of labeling (ProDOL)”. Além disso, foi desenvolvido um pipeline de análise ProDOL para medições de eficiência de marcação por análise de colocalização de molécula única.
A integração dessas técnicas melhora significativamente a capacidade de caracterizar abordagens de marcação fluorescente e medir eficiências de marcação, facilitando a contagem precisa de POIs. Ao identificar condições de marcação ideais para uma contagem robusta de proteínas, é possível aproveitar melhor o potencial da microscopia de fluorescência na pesquisa biológica, levando a uma compreensão mais precisa e confiável dos processos celulares. Esse potencial foi demonstrado na determinação do número de cópias de proteínas em tempo real de SLP-76 em microclusters (MCs) após a ativação do receptor de célula T (TCR) e como esses são afetados pela proteína Nef do vírus da imunodeficiência humana (HIV)-1.
Resultados
ProDOL
ProDOL baseia-se na colocalização dos sinais de moléculas únicas emitidos por etiquetas proteicas marcadas e sinais de referência em imagens espectralmente separadas. Para calcular o DOL, é avaliada a fração de sinal de referência colocalizada com um sinal de etiqueta proteica. Essa estratégia foi implementada criando um constructo de fusão para servir como sonda ProDOL, capaz de avaliar a eficiência de marcação de diferentes etiquetas proteicas em linhas celulares de mamíferos. A sonda ProDOL foi projetada com eGFP como marcador de referência e duas etiquetas proteicas autolabeláveis (HaloTag e SNAP-tag). Uma âncora de membrana derivada da quinase Lyn N-terminal direciona a sonda ProDOL para a membrana plasmática via modificação pós-traducional, permitindo a imagem de molécula única por microscopia de fluorescência de reflexão total interna (TIRF). Um conector α-helicoidal foi adicionado entre SNAP-tag e HaloTag para facilitar a maturação e evitar o dobramento incorreto da proteína de fusão, e uma His-tag C-terminal permite a purificação por afinidade ou imunomarcação.
Com eGFP como marcador de referência, a sonda ProDOL permite a discriminação espectral de substratos SNAP-tag e HaloTag fluorescentes vermelhos e vermelho-distante usados com frequência. Se necessário, eGFP pode ser substituído por proteínas fluorescentes alternativas para facilitar as medições de DOL para substratos de etiquetas proteicas com espectros de excitação ou emissão que se sobrepõem com eGFP. De maneira similar, etiquetas proteicas alternativas podem ser introduzidas na sonda ProDOL.
O nível de expressão da sonda ProDOL é um parâmetro crítico para medições robustas de DOL. Embora uma alta densidade da sonda forneça melhores estatísticas, a densidade deve ser suficientemente baixa para detectar sinais individuais em imagens limitadas por difração. Portanto, a sonda ProDOL foi inserida em um plasmídeo retroviral pBABE para facilitar a integração genômica estável e alcançar níveis de expressão adequados para a localização de sinais de molécula única limitados por difração.
O sinal de fundo inespecífico é outro fator importante que interfere na determinação do DOL em células vivas. Embora eGFP forneça uma etiqueta livre de fundo, a marcação inespecífica tem sido frequentemente observada ao marcar SNAP-tag ou HaloTag em células. Por isso, também foi criada uma versão truncada da sonda ProDOL contendo apenas a âncora de membrana da quinase Lyn e o marcador de referência eGFP, mas sem etiquetas proteicas adicionais (Lyn-eGFP (LynG)). A sonda LynG serve como controle para monitorar a densidade da marcação inespecífica.
Para complementar a sonda, foi criado um fluxo de trabalho de processamento de dados composto por sete módulos que consideram a localização do sinal nas células, aberrações cromáticas, sinais inespecíficos e variações das densidades de marcação. Primeiramente, imagens de moléculas únicas multiplexadas em cores são adquiridas por microscopia TIRF, e uma máscara de segmentação é gerada a partir da imagem do canal de referência para excluir sinais de fundo fora das células da análise subsequente. Os sinais de referência e de marcação são localizados com precisão de subpixel usando o ThunderSTORM e, posteriormente, corrigidos para aberrações cromáticas aplicando uma matriz de transformação afim. Em seguida, a colocalização das etiquetas marcadas com a referência é determinada usando um limite de distância T, no qual a fração de colocalização específica é maximizada enquanto a contribuição da colocalização aleatória é mantida no mínimo. O limite T pode, em teoria, ser definido para o limite de difração. No entanto, aberrações cromáticas, esféricas ou planas não lineares podem levar a um desvio maior que o limite de difração entre funções de distribuição de pontos (PSFs) espectralmente diferentes, tornando benéfico um limite variável T. Os sinais colocalizados são então ajustados para levar em conta os efeitos das variações na densidade de marcação e a presença de sinais inespecíficos.